| 清华张翀团队连续发表两篇重量文章-推进细菌CRISPR筛选新进展 | 您所在的位置:网站首页 › 张翀斌 冬奥会 › 清华张翀团队连续发表两篇重量文章-推进细菌CRISPR筛选新进展 |
清华张翀团队连续发表两篇重量文章-推进细菌CRISPR筛选新进展
|
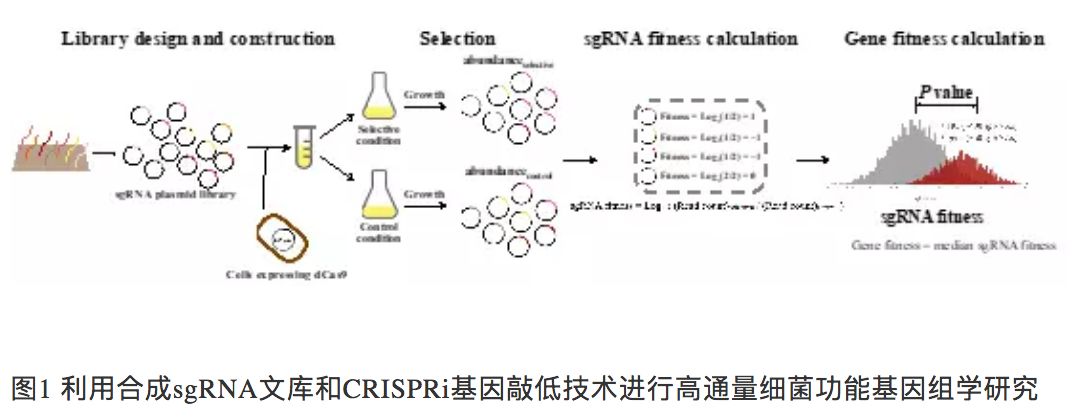
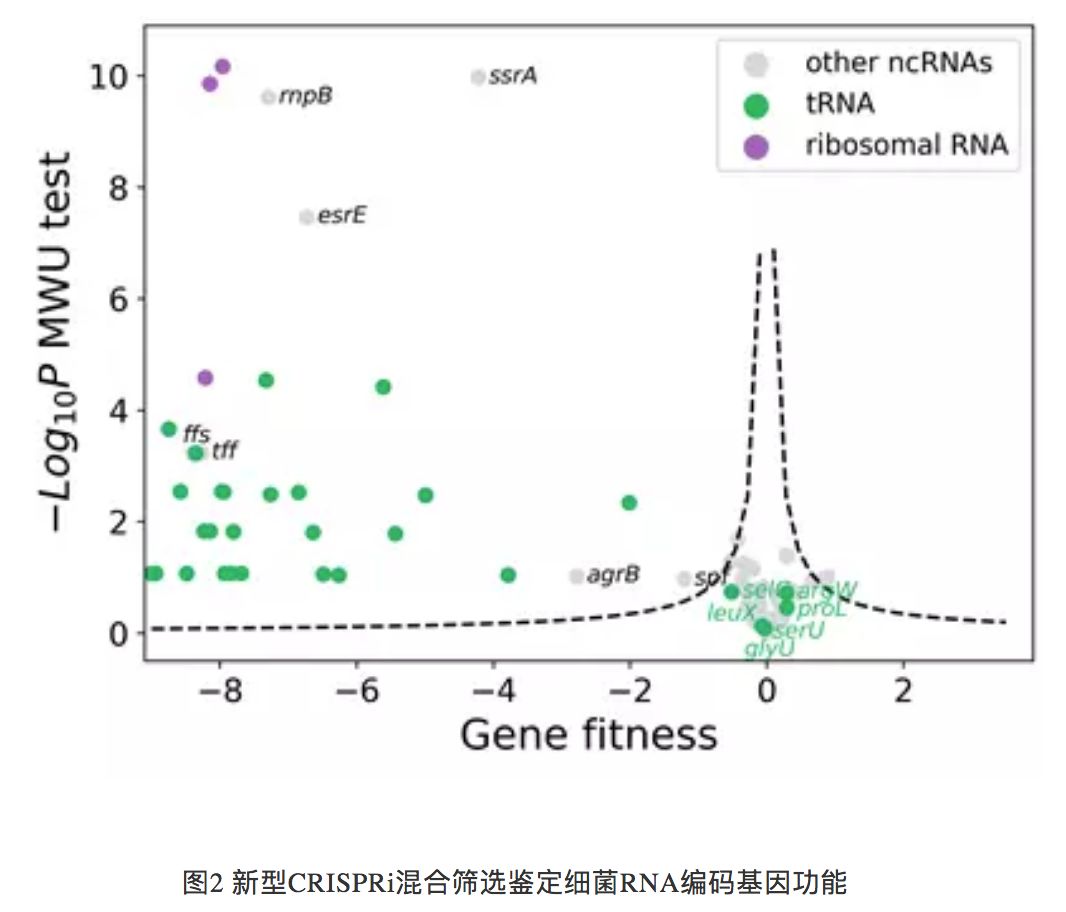
原标题:清华张翀团队连续发表两篇重量文章-推进细菌CRISPR筛选新进展 6月26日,清华大学化工系生物育种技术与装备团队在《自然·通讯》(Nature Communications)发表文章,报道了一种基于CRISPR/dCas9敲低技术(CRISPRi)的新型细菌功能基因组学方法,可以一次性研究细菌中数千个基因和特定表型的关系。
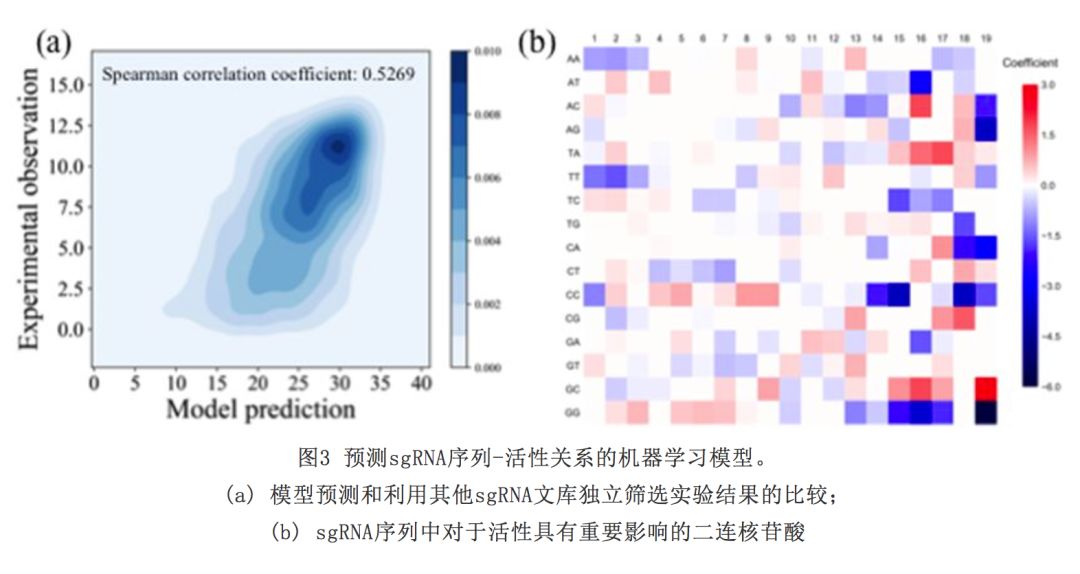
6月30日,清华大学化工系生物育种技术与装备团队在《核酸研究》(Nucleic Acids Research)发表文章,结合高通量筛选技术和机器学习手段,报道了细菌中迄今为止最大规模的sgRNA活性数据集,建立了细菌中第一个利用sgRNA序列特征预测其介导Cas9切割DNA活性的数学模型,用以预测和优化CRISPR/Cas9在细菌基因组编辑中的工作性能。
经过工程化改造的细菌免疫CRISPR系统(Clustered regularlyinterspaced short palindromic repeats)已经发展成为具有广泛应用的基因组编辑工具。近年来,科学家逐渐展示了其在高通量基因功能筛选方面的应用潜力:首先对待筛编码基因进行敲除或敲低,并以细胞生长或特定标记作为一个表型指标,将靶定待研究表型相关基因的单导向RNA(sgRNA)文库富集出来,并利用高通量测序技术定量每个基因的功能。目前这一策略主要被用来进行高等真核生物的高通量功能基因组学研究。由于细菌和高等真核生物基因组的系统性结构差异,在细菌中使用这种方法的高通量功能筛选尚未见报道。在Nature Communications工作中,清华大学研究团队构建了大肠杆菌全基因组范围的sgRNA文库,结合CRISPRi基因敲低技术,对于这一模式微生物的一系列重要基因组学问题开展了大规模研究。 展开全文
同时,在NAR文章中,张翀团队通过机器学习,从而建立起sgRNA序列影响活性的数学模型。该工作获得了迄今为止最大规模的sgRNA活性数据机,建立了细菌第一个利用sgRNA序列特征预测其介导Cas9切割活性的数学模型。 化工系张翀副教授为两篇文章的通讯作者,生物育种技术与装备团队首席邢新会教授,信息国家研究中心、自动化系谢震研究员为本文共同作者,他们同时为清华大学合成与系统生物学研究中心核心成员。NC文章中,清华大学化工系博士生王天民、关长阁为本文的共同第一作者。北京合生基因科技有限公司刘兵亦对此文给予了一定的帮助。该成果得到了国家自然科学基金委重点仪器研发项目、面上项目,国家重点研发计划以及清华大学自主科研计划的资助。NAR文章中,清华大学化工系博士生郭佳荟、王天民为本文的共同第一作者。清华大学计算机系的罗成博士、北京合生基因科技有限公司刘兵亦对此文给予了一定的帮助。该成果得到了国家自然科学基金委重点仪器研发项目、面上项目,国家重点研发计划以及清华大学自主科研计划的资助。 合生基因正面向全球生命科学研究者提供合成生物学科研服务,主要包括哺乳动物细胞基因编辑与调控服务。现已成功为包括清华大学、北京大学、中科院、复旦大学、香港中文大学等科研院所提供包括高通量基因功能筛查与药物抗性筛选服务、CRISPR-Cas9 载体设计与构建服务、shRNA/miRNA 载体设计与构建服务、细胞系工程改造订制服务、gRNA 活性检测试、慢病毒包装、gRNA 文库构建、基因合成服务、基因工程鼠构建在内的科研服务项目。返回搜狐,查看更多 责任编辑: |
【本文地址】